Chapter 4. 变形者的形态(The Shape of a Shapeshifter)
“形式追随功能”是建筑学中的著名格言,但它几乎适用于整个物理世界。锤子和螺丝刀形状各异,各自适应其用途,但两者都有相似的握柄,因为这部分功能——适应人手——是相同的。同样的原理也适用于细胞层面。如果一种蛋白质酶的作用是将食物分解成可代谢的小块,那么该酶就会有一个凹槽来容纳需要分解的食物分子,比如马铃薯淀粉。如果蛋白质的功能是移动肌肉,那么它就需要一个可以伸展和收缩的弹性区域。
形态与功能之间的这种关系意味着,除非我们了解生命分子的结构、它们的构建方式以及如何相互配合,否则我们无法真正理解它们。没有结构信息,生命科学领域的研究人员就像在完全黑暗中试图修理汽车发动机的机械师——过程会非常缓慢、低效且令人沮丧。而掌握结构信息就如同打开了灯;现在机械师可以看到发动机的所有部件、它们如何相互契合、哪个部件或连接存在缺陷,以及如何修复。
第一代分子生物学家将解读蛋白质及随后 DNA 的物理结构视为宏伟而值得挑战的目标。他们采用的技术名为 X 射线晶体学。该方法通过向样本(如蛋白质分子晶体)发射 X 射线束,收集衍射辐射的图像,并逆向推算出产生该衍射的结构。想象一颗石子投入平静的池塘,会产生向外扩散的波纹,这些波纹可用于精确确定石子入水的位置。现在设想将一整把石子撒入池塘,波纹图案会变得复杂交错,但每颗石子落点的信息依然蕴含其中。同理,X 射线束射向蛋白质晶体产生的衍射图案,能够揭示蛋白质中单个原子的位置。
那么 RNA 呢?我们已经见识到 RNA 拥有奇妙的功能,未来还会遇到更多。每种功能必然对应着特定的形态,即实现该功能的具体结构。但事实证明,RNA 的构型比 DNA 更难测定。
吉姆·沃森(Jim Watson)对此深有体会。在共同发现 DNA 双螺旋结构后,沃森曾想再接再厉破解 RNA 的结构。但他遇到了一个难题:DNA 只有一种形态——双螺旋,其中每条链都与另一条配对。这种扭曲的梯状结构将两条链牢牢锁住并相互制约——或者像我们 RNA 科学家常开玩笑说的,它阻止了两条 DNA 链做出任何非常有趣的事情,比如催化作用。而 RNA 则相反,它没有固定的形态,而是有数百万种可能的形状。摆脱了双螺旋的束缚,RNA 几乎可以呈现出无限多种形态,这也解释了它惊人的多功能性。然而,RNA 这种千变万化的特性,使得理解它所采取的各种姿态变得尤为重要。但绘制这种善变物质的结构图一直是个众所周知的难题。
沃森在 RNA 结构研究上挣扎了十年之久。他最初从植物病毒、小牛肝脏和酵母等多种来源中提纯 RNA,进行 X 射线衍射实验,并基于极其粗糙的数据得出结论——这些不同的 RNA 具有单一的共同结构。这就像在浓雾弥漫的天气里,从 200 米外观察一头大象和一辆大众汽车,然后断定它们是相同的。如果你有一副双筒望远镜,并等到太阳出来,就会得出截然不同的结论。
在最终放弃之前,沃森实际上迈出了正确的一步。他从研究功能多样、结构各异的 RNA 混合物转向提纯核糖体——其 RNA 具有特定结构,以适应它们在蛋白质合成中所执行的具体功能。但解析核糖体结构的技术与专业知识还需再发展 40 年才能成熟。
然而,一旦我们终于将复杂的 RNA 结构呈现在眼前,就能直接观察到 RNA 如何施展其魔力——它如何作为机器构建必需的蛋白质分子,如何延伸染色体的末端,或精确编辑人类细胞中的 DNA。所有这些关于 RNA 催化能力的重大发现都还在未来。最初的成功始于从小处着手。
婴儿迈出的第一步
康奈尔大学的鲍勃·霍利【Bob Holley】接过了吉姆·沃森的工作。20 世纪 50 年代末,他意识到试图从混合的 RNA 样本中推测单一结构是徒劳的,因此他将研究重点转向了 tRNA——这种能将氨基酸与其三联体密码子连接的适配分子。转运 RNA 的分子量足够小,使他有机会测定其核苷酸序列——这在当时对任何类型的 RNA 而言都是前所未有的突破。
为什么霍利必须先对 RNA 进行测序,才能尝试解析其结构?解析 RNA 结构有点像给句子画图解。即使你是世界上最伟大的语法学家,如果连句子都读不懂,就别指望能画出图解。核苷酸序列提供了化学碱基 A、U、C、G 的顺序——这些就像句子中的字母——一旦看到它们串联起来,你就能开始绘制分子图解,弄清楚这些元素在空间中的位置以及它们如何协同工作。
作为 tRNA 的来源,霍利选择了酵母——就是我们用来烤面包和酿啤酒的那种酵母。他知道酵母中 tRNA 的含量相对丰富,而且可以从当地面包店买到所需量的弗莱希曼酵母。然而,要纯化出 1 克(约一颗葡萄干的重量)的某一种 tRNA,需要耗费三年时间和 300 磅酵母。
霍利成功分离出的 tRNA 恰好是作为氨基酸丙氨酸适配器的那一种。一旦他和他的研究团队将其分离出来,他们便开始将其切割成足够小的片段以进行化学分析,进而确定这些片段的排列顺序。不到一年时间,他们就破译了核苷酸序列,为解析首个 RNA 结构铺平了道路。
1965 年,鲍勃·霍利团队中经验丰富的研究科学家伊丽莎白·凯勒接受了预测丙氨酸 tRNA 如何在二维空间中自我折叠的挑战。她已知碱基序列,但这些碱基可能如何相互作用呢?
记住 RNA 通常是单链的。在 DNA 中,一条链上的碱基与另一条链上的碱基配对,形成 DNA 梯子的横档,无论序列如何,都会形成相似的双螺旋结构。但对于 RNA 来说,序列决定了形状,因为链的一部分碱基会与另一部分的碱基配对。单个 G-C 配对太弱无法稳定结合,但举个例子,如果有四个连续的 G 可以与四个连续的 C 配对,那么这四个碱基对就能稳固结合。这些由 RNA 序列决定的配对使 RNA 自身折叠,形成“发夹”、分支、环、结以及无数其他可能的形状。凯勒很快意识到,她可以通过多种不同的碱基配对组合来折叠 tRNA。那么,哪一种才是正确的呢?
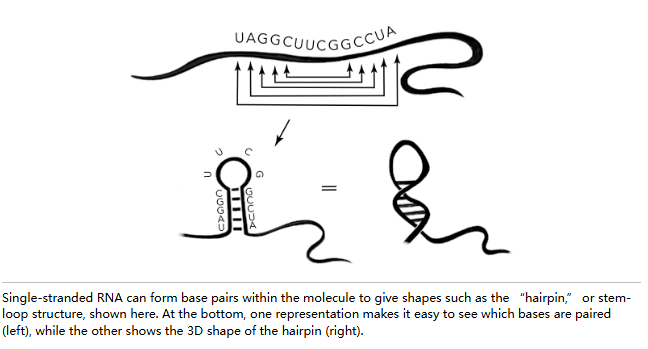
一个线索涉及 tRNA 的三个碱基,它们提供了与 mRNA 密码子的连接。这个三联体与密码子的序列互补,被称为反密码子。凯勒和霍利认为,这个反密码子不会埋藏在 tRNA 结构的折叠中,而是会突出以便与 mRNA 轻松配对,这似乎是合理的。
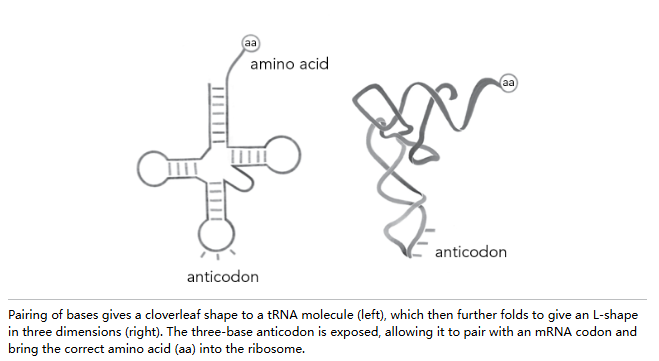
凯勒收集了管道清洁器和魔术贴碎片来模拟不同的碱基配对可能性。她最终确定了一种独特的三叶草模式,这种结构满足了预期:反密码子未配对,位于结构中间臂顶端的环中,随时准备与相应的 mRNA 密码子配对。
很快,其他十几种 tRNA 的序列也被测定出来,理论上每一种都能折叠成三叶草结构。由于所有 tRNA 都需要适配核糖体上的相同位置以递送氨基酸进行蛋白质合成,它们必须具有相同的形状。因此,观察到“一种形状适配所有”现象为 tRNA 的三叶草结构提供了有力支持。
尽管这显然是一个重大突破,但凯勒的三叶草模型有一个很大的缺陷:它平铺在桌面上,仅展示了 tRNA 的二维结构。我有时将这种二维呈现称为“路杀标本”,因为它们展示的 RNA 就像被卡车碾过一样。正如通过分析一只被压扁的松鼠很难理解松鼠的行为一样,人们也无法真正从二维模型中理解 RNA 的行为。
20 世纪 60 年代末,一场揭示 tRNA 三维结构的竞赛爆发了。这是剑桥与剑桥之间的对决:一支团队由麻省理工学院的 Sung-Hou Kim 和 Alex Rich 领导,另一支则由分子生物学实验室的 J. D. Robertus 和 Aaron Klug 领衔,后者位于另一个剑桥。
首选方法是 X 射线晶体学,当时这种方法正快速解析出各种蛋白质结构。在实验室中培养晶体需要制备高度浓缩的纯化分子溶液,然后排列一系列含有不同浓度添加剂(如盐类)的液滴。我们寻找的是能形成合适晶体的"恰到好处"条件。如果分子在某个液滴中溶解度过高,溶液会保持澄清——无法形成晶体;若在另一液滴中溶解度过低,分子会从溶液中沉淀成无用的团块。但偶尔某个液滴的溶液恰好处于溶解与不溶解的临界点时,分子就会整齐排列成行与列。通过显微镜观察,你会看到边缘锐利的美丽晶体缓慢生长,日渐增大。随后将单颗晶体置于 X 射线束前,开启辐射并收集衍射图案。经过更多技巧处理和大量计算后,就能"解析"出结构——即获得分子中每个原子在三维空间中的位置模型。
结晶既是科学也是艺术,因此研究人员学会了接受所能得到的结果。最容易结晶的是苯丙氨酸 tRNA,所以两个竞争团队都解析了它的结构。其结构表明,伊丽莎白·凯勒设想的三叶草结构确实存在,且三叶草进一步自我折叠形成了 L 形分子。在“L”形的一端是三联体反密码子,另一端则是对应的苯丙氨酸氨基酸。
确定首个具有已知生物功能的 RNA 三维结构是一项激动人心的成就——1974 年由剑桥的两个团队共同实现。这类情况下,首次突破往往会在短期内引发一系列后续发现。第一块多米诺骨牌倒下,引发连锁反应。但 RNA 结构领域却并非如此。在苯丙氨酸 tRNA 之后的 15 年里,再没有比 tRNA 更大的 RNA 结构被解析。尽管无数研究者付出了巨大努力,其他所有已知类型的 RNA 都像泥鳅般难以捕捉。由于缺乏结构作为指导,要弄清大型 RNA(如四膜虫核酶)中单个核苷酸的作用,就成了一个极其缓慢而乏味的过程。
蝴蝶捕手
当一些人还在等待技术足够成熟以结晶更复杂的 RNA 类型时,弗朗索瓦·米歇尔已经开始梦想了。弗朗索瓦是法国国家科学研究中心(CNRS)位于巴黎郊外吉夫-叙尔-伊维特镇的一名研究员。他的一大爱好是收集、培育和研究不同种类蝴蝶的遗传基础,同时他也热衷于收集 RNA 序列。他对这些序列的记忆力惊人。据说他会在脑海中比较它们,并尝试以各种方式将它们组合在一起,甚至在睡觉时也是如此。他看起来就像个古怪的天才——浓密的头发,大胡子。当我在会议上见到他时,有时会以为他是刚从森林里经过数月的蝴蝶搜寻后出来的。
弗朗索瓦在吉夫的同事们发现,酵母线粒体(细胞的能量生成部分)中存在一组新的内含子,这些内含子具有令人着迷的遗传特性。他们注意到,几乎相同的短小序列片段散布在九个不同的内含子中。从一个酵母内含子到另一个,这些片段以相同的顺序排列,暗示它们具有相似的功能。弗朗索瓦知道这些相似的序列片段对 RNA 剪接反应至关重要,因为当这些 RNA 片段发生突变时,剪接反应就无法进行。此外,这些序列片段成对互补,表明它们可以像拉链一样结合形成茎环结构(如上图所示)。到 1982 年,弗朗索瓦提出了这些配对的 RNA 序列如何能为所有这些酵母线粒体内含子形成相似的二维结构。
但内含子为何需要形成特定的形状呢?毕竟,如果它们类似于菲尔·夏普和里奇·罗伯茨发现的 mRNA 内含子,内含子 RNA 可能需要保持非结构化才能与 U1 和 U2 snRNA 配对。然而,结构化内含子的谜团并未持续太久——就在弗朗索瓦提出他的结构模型后不久,我们报道了在四膜虫中发现核酶的消息。弗朗索瓦仅用了一秒钟观察四膜虫内含子的核苷酸序列,就意识到他的二维模型同样适用于我们的自剪接内含子。这既惊人又出乎意料:酵母在进化上与纤毛原生生物(如四膜虫)相距甚远,线粒体基因也与细胞核基因截然不同。那么这些毫无关联的 RNA 为何会折叠成相同形状?前所未闻的答案必然是——这种形状对自剪接催化剂不可或缺,而酵母线粒体内含子必然也能自剪接。果然,1985 年荷兰团队证实了这一预言。
弗朗索瓦的二维模型看起来有点像 tRNA 三叶草结构的扩展版本。内含子 RNA 比 tRNA 大几倍,其结构也更为复杂,拥有十几个甚至更多的茎环结构,而 tRNA 只有四个。掌握这类核酶的二维结构图是一项重大成就,但弗朗索瓦深知 RNA 的催化作用并非在二维空间进行。他渴望仅凭序列构建完整的 3D 模型,这是此前从未有人为大型 RNA 实现过的壮举。
1983 年,弗朗索瓦在一次科学会议上结识了斯特拉斯堡大学的埃里克·韦斯特霍夫。埃里克童年时期在比属刚果度过,在比利时列日大学获得物理学学位,后于威斯康星大学通过 X 射线晶体学技术研究 tRNA 结构。埃里克的学术背景与专业技能与弗朗索瓦完美互补,两人都热衷于将 RNA 的二维模型转化为三维现实。
斯特拉斯堡位于葡萄园遍布的莱茵河谷,离巴黎并不很近,因此弗朗索瓦来找埃里克进行模型构建时总会带上睡袋。埃里克坐在电脑前,构建着与四膜虫内含子结构中已知碱基配对区域对应的 RNA 螺旋。这部分并不困难——RNA 双螺旋就像 DNA 双螺旋的小片段,其螺旋角度的细节遵循 tRNA 晶体结构中观察到的规律。真正的难点在于弄清楚这些小螺旋单元如何在三维空间中组合成催化形状。但愿连接这些螺旋的 RNA 序列能为它们的立体排布提供线索,就像这些连接序列将 tRNA 的三叶草结构折叠成 L 形三维结构那样。
弗朗索瓦会坐在埃里克旁边,仔细研究 87 个相关内含子的打印序列,这些都属于自剪接类型。这些打印资料补充了他脑海中储存的大量序列。当埃里克在电脑上重新排列 RNA 片段时,弗朗索瓦会观察不同内含子序列间的协同变化,这些变化暗示了某些核苷酸在三维空间中的相互接触。每当发现此类证据,他就会宣布:"这个成立!"夜晚,他蜷缩在埃里克电脑旁的睡袋里,脑海中 RNA 序列的影像如蝴蝶般翩翩起舞。
到 1990 年,弗朗索瓦和埃里克已经构建出了四膜虫内含子的三维模型——在 RNA 生物学家眼中,这简直美不胜收。它看起来就像一个被父母环抱的婴儿。"婴儿"是一段含有需要被切割和剪接位点的 RNA 螺旋。其中一位"家长"是弗朗索瓦早先证实能定位必需鸟苷的 RNA 结构部分,这个鸟苷就像"剪刀"一样负责剪除内含子。另一位被称为 P4-P6(配对区域 4-6)的"家长",则负责支撑这些关键 RNA 元件的精确定位。
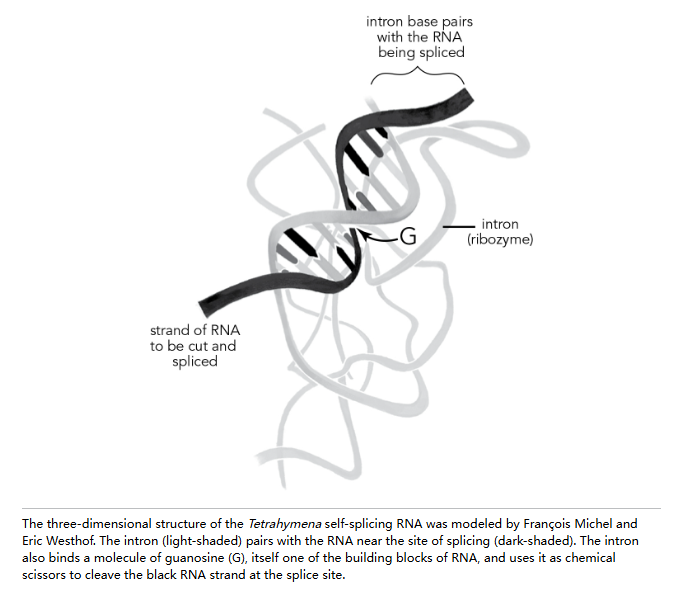
模型完美地契合在一起,但它与真实结构有多接近?要找出答案,需要 X 射线晶体学以及一位来自夏威夷的年轻女性的才华和毅力。
让我们一目了然
詹妮弗·杜德娜在夏威夷大岛葱郁的东海岸长大。她从小就被当地潮汐池的奇观和沿着活跃的基拉韦厄火山口徒步的震撼所吸引,早早迷上了科学。她横跨太平洋来到加州波莫纳学院攻读生物化学本科,随后又横穿美国前往哈佛医学院攻读博士学位。在那里,她的论文研究聚焦于四膜虫核酶的功能。
这让我们多少成了竞争对手,但竞争始终是友好的。有一次,在她研究生学习期间,珍妮弗来博尔德拜访我,这位纤弱的女子令人无法不感到钦佩(甚至略带敬畏)。她拥有一种不可思议的天赋,能设计出恰好验证任何假设的实验,而且她所展现出的精力与干劲,远超我见过的任何科学家。因此,当她在 1989 年获得博士学位并请求加入我们在博尔德的博士后研究时,我立刻答应了。
珍妮弗和我,以及 RNA 研究领域的许多同行都坚信,要理解任何核酶的工作原理,必须获得其三维结构图像。但 RNA 结构研究当时正遭遇瓶颈——自 tRNA 结构破解后的 15 年间,再无大型 RNA 结构被揭示。我们知道,获取这样的图像是一项雄心勃勃的任务。然而,若能成功,这将成为一项里程碑式的成就,注定会被载入全球教科书。
核酶结构有望成为解答 RNA 结构基本问题的金矿,包括 RNA 如何折叠形成特定催化结构这一著名谜题。蛋白质折叠及形成酶活性位点的方式已广为人知:蛋白质酶将所有疏水侧链包裹在内,形成疏水(厌水)核心,在其亲水(亲水)外表支撑起一个催化活性裂隙。但 RNA 不可能运用相同原理构建结构,因为它根本不具备任何可用的疏水单元。更棘手的是,RNA 链的每个环节都带负电荷,而蛋白质链大多不带电。#要让 RNA 形成紧密结构,就意味着要让所有这些负电荷聚集在一起——这就像把一堆磁铁的南极都朝内摆放后试图压合它们,结果只会相互排斥。tRNA 结构仅为 RNA 结构提供了一种范式,且不具备催化功能;在核糖体和 mRNA 缺失的情况下,它自身完全无法发挥作用。 四膜虫核酶的结构将首次揭示一个大 RNA 分子如何能像蛋白质那样折叠,尽管表面上看起来缺乏这样做的条件。
1991 年詹妮弗抵达博尔德时,我们一致认为尝试解析整个四膜虫核酶的结构可能过于雄心勃勃——这个 414 碱基的分子比 tRNA 大了约六倍。于是我们决定先以该分子的半部分作为 X 射线结晶目标,但并非随意选取一半,而是必须选择在功能和结构上都值得深入研究的部分。我实验室的研究生费莉西亚·墨菲已鉴定出一个名为“P4-P6”的关键区域符合要求。她发现 P4-P6 会像老式木制衣夹那样自我折叠,对于精确定位 RNA 中含有两个剪接位点之一的部分至关重要——尽管原子层面的细节尚不明确。揭示这些细节将成为詹妮弗的任务。
詹妮弗与我的实验室的科学家安妮·古丁合作,很快她们就开始合成 P4-P6 RNA,并在一系列盐条件下设置结晶滴。不久后,她们找到了一种配方,能够稳定地产生边缘清晰美丽的晶体。推测 RNA 分子在晶体中排列成整齐的行和列,但最初这些晶体在 X 射线束下产生的衍射图案并不理想。辐射破坏了晶体中的 RNA,导致无法获得清晰的图像。
1993 年,作为 RiboSki 团队中坚力量的两位耶鲁大学教授琼和汤姆·施泰茨正在博尔德进行为期一年的学术休假。他们访问了我的研究小组以及同事奥尔克·乌伦贝克的实验室。汤姆·施泰茨是全球顶尖的 X 射线晶体学家之一,他在耶鲁的团队曾解析出包括关键 RNA-蛋白质和 DNA-蛋白质复合物在内的重要结构。汤姆喜欢在我们的休息室讨论科学,有次与詹妮弗交谈时,他提到自己的团队开始采用冷冻晶体技术来减少 X 射线损伤——他们用液氮将液态丙烷冷却至极低温度,再将晶体快速浸入液态丙烷中急速冷冻,从而避免冰晶形成。詹妮弗和安妮掌握了这项技术后欣喜地发现,P4-P6 RNA 的 X 射线衍射图谱质量显著提升,其清晰度足以让我们定位折叠 RNA 结构中单个原子的位置。
但通常情况下,科研进展总是进两步退一步——这还算顺利的时候。我们遭遇的重大挫折是一个技术难题,这个问题在詹妮弗于博尔德期间一直困扰着我们。计算结构不仅需要你研究的分子晶体,还需要它的“重原子衍生物”——即分子中嵌入一个或多个固定位置的重原子。所谓“重原子”是指质子、中子和电子数量较多的原子,如铂、金、银、汞、硒、钨或锇。只有通过比较原始分子与重原子衍生物的衍射图案,才能计算出分子的三维结构。虽然蛋白质口袋中嵌入重原子的方法已广为人知,但 RNA 却截然不同,那些对蛋白质有效的重原子对 RNA 完全不起作用。
因此,P4-P6 结构暂时仍未破解。然而,在博尔德工作三年后,詹妮弗凭借她在哈佛的博士论文研究以及在博尔德取得突破性 RNA 结构研究的进展,赢得了极高的声誉,各大学纷纷争相聘请她。她选择了耶鲁大学——很大程度上是因为她与汤姆·施泰茨和琼·施泰茨已建立的深厚关系。她带上了博尔德的研究生杰米·凯特,后者继续尝试用各种金属作为重原子衍生物进行实验。在多次失败后,杰米发现锇离子的大小恰好可以替代折叠 RNA 中的镁离子。一种合适的锇化合物由斯坦福大学的一位即将退休的化学家合成。幸运的是,杰米在他的实验室清理前联系上了他,并获得了这种后来被证明是“神奇金属”的化合物。锇化合物确实替代了 RNA 中三个特定位置结合的镁离子,使得杰米和詹妮弗最终在 1996 年成功解析了 P4-P6 RNA 结构。
这个结构令人叹为观止。它揭示了 RNA 分子如何能够自我折叠形成一个紧密的内部核心,这是蛋白质常见的特性,但对 RNA 来说似乎极具挑战性。然而,对于一个催化 RNA 来说,它形成类似蛋白质的结构是有道理的——RNA 表现得像蛋白质,那么为什么它看起来不应该也像蛋白质呢?该结构还展示了带正电荷的镁离子(活细胞的正常成分)如何定位自身以解决高度带负电的 RNA 折叠时的电荷排斥问题。再次联想到磁铁,如果你想将两块磁铁的负极靠在一起并保持这种状态,只需在它们之间放置一个正极即可。
下一步是解决整个核酶的结构。Barb Golden 来到博尔德进行博士后研究,并于 1998 年在 RNA 晶体学方面创下了新的尺寸记录:一个 247 核苷酸版本的嗜热四膜虫内含子,具有生物催化活性。这段 RNA 包含了 P4-P6 结构域,正如我们所预测的那样,活性核酶中的 P4-P6 结构域与单独存在的 P4-P6 结构域看起来几乎一模一样。Barb 的内含子结构还显示出一个由 RNA 形成的“摇篮”,等待着拥抱含有剪接位点的 RNA 螺旋;它与 Michel 和 Westhof 八年前预测的结果惊人地相似,令人满意。
破解四膜虫 P4-P6 区域的结构激发了 RNA 结构这一小领域的研究热潮,很快其他大型功能性 RNA 分子的晶体结构也相继被解析。这些结构作为一个整体,展现了 RNA 的巨大潜力。每种功能性 RNA 自然有其独特形态,但四膜虫核酶中揭示的普遍规律注定会在众多其他 RNA 中重现。这一结构完美诠释了仅凭 A、G、C、U 四种碱基就能构建何等复杂的分子机器。
大约在接下来的十年里,RNA 结构领域的研究基本处于停滞状态。每年仅能解析出一两种新结构,科学家们虽在稳步前进,但与已解析的数万种蛋白质结构相比,RNA 专属结构的报道数量不足其百分之一。这种局面令人沮丧——它不仅阻碍了我们对 RNA 基础认知的深入,更延缓了可能拯救生命的医疗创新进程。研发抗病药物的工业科学家需要目标分子的精细结构图来指导研究:针对蛋白质靶点,他们通常能查阅他人已解析的结构,或基于庞大相似结构数据库进行计算推演;但对于大多数 RNA 靶点,其结构的未知性直接制约了药物开发。我们亟需一场 RNA 结构解析的范式革命。
群体的智慧
预测 RNA 结构不止一种方法,可以避开耗时且不确定的 X 射线晶体学过程。我们已经见识过一种途径:找到像弗朗索瓦·米歇尔和埃里克·韦斯特霍夫这样对 RNA 折叠原理有深刻见解的聪明人,并给他们几年时间来解决这个问题。但反过来呢——召集数千名毫无 RNA 结构经验的非科学家,每人花几个小时来攻克这个难题?我们都听说过众包。这种方法能在 RNA 折叠这样深奥的领域奏效吗?又会有多少人愿意参与其中?
2017 年 1 月,我坐在斯坦福大学里朱·达斯的办公室里,惊讶得合不拢嘴。当一位同行科学家告诉你他或她完成了一件你根本无法想象的壮举时,那种震撼总是令人谦卑。里朱让来自全球的 37,000 人玩一款名为 EteRNA 的电脑游戏,而这些玩家正在为 RNA 折叠问题寻找解决方案。
2009 年,eteRNA 在网上宣布了它的第一个挑战:设计一种能折叠成五角星或十字架形状的 RNA。换句话说,怎样的 A、G、C、U 序列才能形成正确的碱基配对,从而折叠成目标形状?参与者来自各行各业。有些是从事 RNA 研究的研究生,有些则是几乎没听说过 RNA 但热衷于尝试新谜题的数独爱好者。有人开发了折叠 RNA 的计算机程序,也有人坚持用纸笔计算。玩家们将答案提交到 eteRNA 网站,然后大家投票选出最有可能折叠成目标形状而非其他形状的序列。得票数最高的八个序列随后在斯坦福大学被实际合成出来。每个序列都通过一种名为 SHAPE 的巧妙方法进行测试,该方法由我实验室的前博士后、现北卡罗来纳大学教堂山分校教授凯文·威克斯发明。SHAPE 技术涉及用仅与单链核苷酸反应的化合物处理 RNA,然后识别哪些核苷酸具有反应活性。 例如,如果一段 RNA 序列确实折叠成五角星形状,它应该具有如下所示的特征性 SHAPE 反应模式。
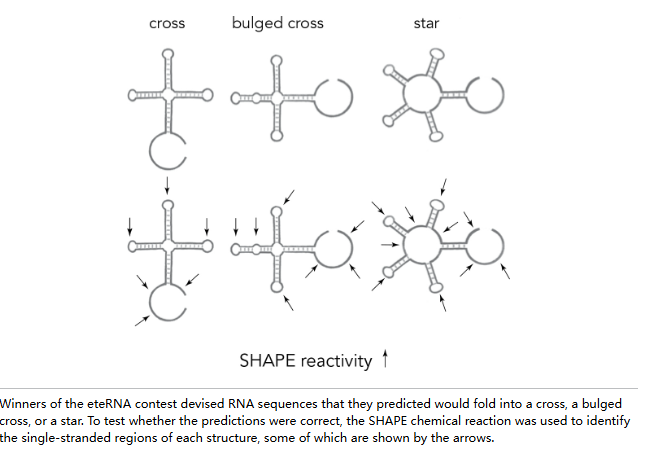
三万七千名玩家接受了挑战,其中顶尖玩家成功解决了每一道难题。成果之显著,甚至催生了一篇极为罕见的论文——该论文的合著者名单中赫然列有 100 位 eRNA 游戏玩家。截至 2022 年,玩家群体已破解了 4,181,632 个 RNA 结构谜题,其中多数玩家在沉迷游戏前对 RNA 几乎一无所知。
近年来,eteRNA 提高了挑战难度,着手解决实际研究问题。例如,在 2020 年的"开放疫苗"竞赛中,玩家们竞相设计一种无需超低温储存的改进版新冠 mRNA 疫苗。其科学假设——更准确地说,是基于专业知识的推测——是:若设计的 mRNA 既能折叠成高度碱基配对的结构,又能保留编码冠状病毒刺突蛋白的能力,将提升 mRNA 在储存期间及注入人体后的稳定性。由于许多氨基酸由两个、四个甚至六个密码子编码,理论上存在数量惊人的可编码刺突蛋白的序列——这个数字(10 的 630 次方)近乎无限,任何计算机都无法遍历如此庞大的序列组合。于是 eteRNA 将这个问题众包给任何愿意参与的游戏玩家,静候解决方案涌现。由于竞赛启动于 2020 年 3 月 18 日,正值新冠疫情导致许多人居家隔离期间,参与人数异常踊跃。
游戏玩家们设计出了许多 mRNA 序列,这些序列堪称真正的“超级折叠者”,大部分碱基都被锁定在碱基对中。但它们实际上更稳定吗?在斯坦福大学,研究人员对其中八种超级折叠 RNA 在储存期间以及进入人体细胞后的稳定性进行了测试。令人欣慰的是,这些序列的稳定性比当前计算机程序设计的序列高出两倍。此刻斯坦福研究团队屏息以待:这些 RNA 是否因折叠过于紧密而无法通过核糖体合成刺突蛋白?无需担心,从 mRNA 到蛋白质的翻译过程完全正常。最终,他们将超级折叠 mRNA 交给辉瑞疫苗团队,在疫苗配方中测试其持久性。在高温环境下存放两周后,采用超级折叠技术的疫苗大部分保持完整,表现远超现有技术设计的 mRNA 疫苗。 由于已获批的新冠 mRNA 疫苗需要在超低温条件下储存和运输,这给向贫困国家配送带来了挑战,因此这种增强的热稳定性具有令人振奋的潜力,有朝一日可能成为更易获取且更廉价疫苗的基础。
人工智能来救援?
利用群体智慧是解析 RNA 结构的一种独特方法。但该领域的未来更可能依赖于用机器学习取代人脑。人工智能(AI)已经能够撰写新闻稿件和社交媒体帖子;它能将口语转化为文字;理论上,它还能让自动驾驶汽车安全地在城市中穿行。那么,我们能否放手让它预测 RNA 结构呢?它能否为米歇尔和韦斯特霍夫省下预测自剪接内含子结构所花费的七年时间?能否为我的实验室节省通过 X 射线晶体学解析自剪接内含子结构所耗费的七年光阴?能否让 37,000 人免于玩 eteRNA 游戏来寻找折叠成星形或十字形的最佳序列?答案几乎肯定是“可以”,尽管目前尚未实现,但未来方向已然明朗。
2021 年,eteRNA 联合创始人 Rhiju Das 与其斯坦福大学同事 Ron Dror 宣布了一项突破。他们成功设计出一款人工智能计算机程序,能较准确地预测给定 RNA 序列折叠时会呈现的 3D 结构。他们面临的挑战之一是构建足够的"训练集"——AI 程序需要先学习真实数据才能探索未知领域。区分猫狗图片对 AI 而言轻而易举,因为网络上有数百万张标注"狗"或"猫"的图片可供训练。但在 RNA 折叠领域,Das 和 Dror 仅掌握 18 组序列与结构配对的训练数据。更困难的是,他们要求 AI 不仅要识别 RNA 结构,还要根据 A、G、C、U 核苷酸序列预测正确的 3D 构型。令人惊叹的是,仅凭这 18 组训练序列,他们的程序就超越了以往的所有结构预测方法。
如何衡量 RNA 三维结构预测的成功与否?埃里克·韦斯特霍夫邀请了全球 RNA 结构生物学家参与他称之为"RNA-Puzzles"的趣味竞赛。当某位参与科学家通过 X 射线晶体学等方法解析出新 RNA 结构时,会暂缓公开结果,给予其他玩家一个月时间仅凭核苷酸序列来预测该结构。简而言之,玩家们处于"答案盲测"状态。部分玩家开发了自动化网络服务器进行结构预测,另一些则采用手工建模方式。所有预测结果汇总后,截止日期一到便会揭晓真实结构。
在四次 RNA-Puzzles 挑战中,Das-Dror 人工智能方法提交的模型在所有参与者中准确度最高。虽然尚未完美,但它正稳步成为科研界值得信赖的工具。可以预见,未来这类结构问题无需踏入实验室即可解决——这对科学进步而言令人振奋,但对于我们这些毕生在实验室里调配分子配方并品味成果的人来说,却难免有些伤感。
三维视角下的 tRNA 与核酶对 RNA 科学产生了深远影响。科学家们得以观察 tRNA 如何折叠以适应核糖体结构——它们不仅携带正确的氨基酸,还精准引导这些氨基酸相互靠近以促进反应,构建蛋白质链。通过原子级细节,人们看清了四膜虫核酶如何定位鸟苷攻击 RNA 剪接位点,RNase P 如何催化特定键断裂以生成成熟 tRNA,以及 snRNAs 如何精密调控 mRNA 的剪接过程。
除了单纯理解生物学运作机制外,掌握 RNA 结构还能让科学家和生物工程师设计结构变体,将其重新应用于新用途。例如,生物工程师可能设计出一种核酶,作为分子电路的一部分用于检测环境样本中的有毒化合物或特定病毒。如今整个合成生物学领域都在使用核酶作为传感器和开关,但这一切都需以已知 RNA 结构为起点。
在测定越来越大的 RNA 三维结构方面取得的突破,激励着该领域的研究者们提升他们的抱负。一些勇敢的科学家甚至开始将目光投向细胞的最后疆界——那个长期保持能量来源之谜的分子机器之母。他们的目标是解析核糖体的结构。
#:在蛋白质中发现的 20 种氨基酸中,15 种不带电荷,2 种带负电荷,2 种带正电荷,而组氨酸(histidine)具有一种非常有用的特性:当环境变得更酸性时,它会从不带电荷转变为带正电荷。